Topológia becslő eljárások
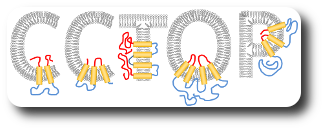
 A csoportvezető a 90-es évek vége óta foglalkozik transzmembrán fehérjék szekvencia analízisével és jelentős eredményeket ért el ezen fehérjék topológia becslése területén. A legújabb kutatásokban olyan eljárást dolgoztunk ki, amelyben egyrészt figyelembe vesszük a jelenleg legnagyobb pontossággal becslő algoritmusok becslését, másrészt olyan topológiai adatokat, amelyeket kísérletes eszközökkel határoztak meg, vagy valamilyen bioinformatika megfigyelés eredménye. A kidolgozott konszenzus, megszorításokat alkalmazó eljárás (CCTOP) az eddigi legpontosabb topológia becslő eljárás, amelyet felhasználtunk az emberi transzmembrán proteom (HTP adatbázis) létrehozásában is.
A csoportvezető a 90-es évek vége óta foglalkozik transzmembrán fehérjék szekvencia analízisével és jelentős eredményeket ért el ezen fehérjék topológia becslése területén. A legújabb kutatásokban olyan eljárást dolgoztunk ki, amelyben egyrészt figyelembe vesszük a jelenleg legnagyobb pontossággal becslő algoritmusok becslését, másrészt olyan topológiai adatokat, amelyeket kísérletes eszközökkel határoztak meg, vagy valamilyen bioinformatika megfigyelés eredménye. A kidolgozott konszenzus, megszorításokat alkalmazó eljárás (CCTOP) az eddigi legpontosabb topológia becslő eljárás, amelyet felhasználtunk az emberi transzmembrán proteom (HTP adatbázis) létrehozásában is.
Témához kapcsolodó közlemények
-
Dobson L, Szekeres LI, Gedán C, Langó T, Zeke A és Tusnády GE (2023) TmAlphaFold database: membrane localization and evaluation of AlphaFold2 predicted alpha-helical transmembrane protein structures Nucleic Acids Research , .
-
Bakos É, Németh O, Kucsma N, Tőkési N, Stieger B, Rushing E, Tőkés A, Kele P, Tusnády GE és Özvegy-Laczka Cs (2022) Cloning and characterization of a novel functional organic anion transporting polypeptide 3A1 isoform highly expressed in the human brain and testis Frontiers in Pharmacology 13, 958023.
-
Dobson L és Tusnády GE (2021) MemDis: Predicting Disordered Regions in Transmembrane Proteins Int. J. Mol. Sci. 22, 12270.
-
Czimer D, Porok K, Csete D, Gyüre Zs, Lavró V, Fülöp K, Chen Z, Gyergyák H, Tusnády GE, Burgess SM, Mócsai A, Váradi A és Varga M (2021) A New Zebrafish Model for Pseudoxanthoma Elasticum Front Cell Dev Biol 9, 628699.
-
Dobson L, Zeke A és Tusnády GE (2021) PolarProtPred: Predicting apical and basolateral localization of transmembrane proteins using putative short linear motifs and deep learning. Bioinformatics , btab480.
-
Zeke A, Dobson L, Szekeres LI, Langó T és Tusnády GE (2021) PolarProtDb: A Database of Transmembrane and Secreted Proteins showing Apical-Basal Polarity J Mol Biol 433, 166705.
-
Kozák E, Szikora B, Iliás A, Jani PK, Hegyi Z, Matula Zs, Dedinszki D, Tőkési N, Fülöp K, Pomozi V, Várady Gy, Bakos É, Tusnády GE, Kacskovics I és Váradi A (2020) Creation of the first monoclonal antibody recognizing an extracellular epitope of hABCC6 FEBS Lett 595, 789-798.
-
Bakos É, Tusnády GE, Német O, Patik I, Magyar C, Németh K, Kele P és Özvegy- Laczka C (2020) Synergistic transport of a fluorescent coumarin probe marks coumarins as pharmacological modulators of Organic anion-transporting polypeptide, OATP3A1 Biochem Pharmacol. , .
-
Langó T, Pataki ZG, Turiák L, Ács A, Varga JK, Várady, G, Kucsma, N, Drahos, L és Tusnády GE (2020) Partial Proteolysis Improves the Identification of the Extracellular Segments of Transmembrane Proteins by Surface Biotinylation Scientific Reports 10, 8880.
-
Vargja, JK és Tusnady, GE (2019) The TMCrys server for supporting crystallization of transmembrane proteins. Bioinformatics 35, 4203-4.
-
Muller, A, Lango, T, Turiak, L, Acs, A, Varady, G, Kucsma, N, Drahos, L és Tusnady, GE (2019) Covalently modified carboxyl side chains on cell surface leads to a novel method toward topology analysis of transmembrane proteins Scientific Reports 9, 15729.
-
Varga J és Tusnady, GE (2018) TMCrys: predict propensity of success for transmembrane protein crystallization. Bioinformatics , .
-
Lango T, Rona G, Hunyadi-Gulyas E, Turiak L, Varga J, Dobson L, Varady Gy, Drahos L, Vertessy BG, Medzihradszky FK, Szakacs G és Tusnady GE (2017) Identification of extracellular segments by mass spectrometry improves topology prediction of transmembrane proteins Scientific Reports 7, 42610.
-
Varga J, Dobson L, Remenyi I és Tusnady GE (2017) TSTMP: target selection for structural genomics of human transmembrane proteins. Nucleic Acids Res 45, D325-330.
-
Varga J, Dobson L és Tusnady GE (2016) TOPDOM: database of conservatively located domains and motifs in proteins. Bioinformatics 32, 2725-2726.
-
Dobson L, Remenyi I és Tusnady GE (2015) The human transmembrane proteome. Biol Direct 10, 31.
-
Dobson L, Remenyi I és Tusnady GE (2015) CCTOP: a Consensus Constrained TOPology prediction web server. Nucleic Acids Res 43, W408-12.
-
Kiss K, Kucsma N, Brozik A, Tusnady GE, Bergam P, van Niel G és Szakacs G (2015) Role of the N-terminal transmembrane domain in the endo-lysosomal targeting and Biochem J 467, 127-39.
-
Dobson L, Lango T, Remenyi I és Tusnady GE (2015) Expediting topology data gathering for the TOPDB database. Nucleic Acids Res 43, D283-9.
-
Tusnady GE és Simon I (2010) Topology prediction of helical transmembrane proteins: how far have we reached? Curr Protein Pept Sci 11, 550-61.
-
Tusnady GE, Kalmar L, Hegyi H, Tompa P és Simon I (2008) TOPDOM: database of domains and motifs with conservative location in Bioinformatics 24, 1469-70.
-
Tusnady GE, Kalmar L és Simon I (2008) TOPDB: topology data bank of transmembrane proteins. Nucleic Acids Res 36, D234-9.


 A csoportvezető a 90-es évek vége óta foglalkozik transzmembrán fehérjék szekvencia analízisével és jelentős eredményeket ért el ezen fehérjék topológia becslése területén. A legújabb kutatásokban olyan eljárást dolgoztunk ki, amelyben egyrészt figyelembe vesszük a jelenleg legnagyobb pontossággal becslő algoritmusok becslését, másrészt olyan topológiai adatokat, amelyeket kísérletes eszközökkel határoztak meg, vagy valamilyen bioinformatika megfigyelés eredménye. A kidolgozott konszenzus, megszorításokat alkalmazó eljárás (CCTOP) az eddigi legpontosabb topológia becslő eljárás, amelyet felhasználtunk az emberi transzmembrán proteom (HTP adatbázis) létrehozásában is.
A csoportvezető a 90-es évek vége óta foglalkozik transzmembrán fehérjék szekvencia analízisével és jelentős eredményeket ért el ezen fehérjék topológia becslése területén. A legújabb kutatásokban olyan eljárást dolgoztunk ki, amelyben egyrészt figyelembe vesszük a jelenleg legnagyobb pontossággal becslő algoritmusok becslését, másrészt olyan topológiai adatokat, amelyeket kísérletes eszközökkel határoztak meg, vagy valamilyen bioinformatika megfigyelés eredménye. A kidolgozott konszenzus, megszorításokat alkalmazó eljárás (CCTOP) az eddigi legpontosabb topológia becslő eljárás, amelyet felhasználtunk az emberi transzmembrán proteom (HTP adatbázis) létrehozásában is.

